NEUROPATHOLOGY AND APPLIED NEUROBIOLOGY:高通量单细胞RNA表达分析方法识别人类周细胞标记
时间:2023-10-21 17:37:20 热度:37.1℃ 作者:网络
周细胞存在于大脑和其他组织的血管壁上,在健康和疾病状态中起着关键作用。例如,周细胞在血流调节、神经炎症、血管生成和介导其他脑细胞类型(如星形胶质细胞和内皮细胞)之间的功能相互作用中发挥重要作用。周细胞功能障碍现在被认为是血管疾病和神经退行性疾病进展的潜在因素,特别是在老年人中。尽管关于周细胞生物学的科学文献迅速发展,但关于这种迷人的细胞类型还有很多有待学习的地方。

动物模型是研究脑周细胞的有效实验环境。对啮齿动物的研究特别有价值,并对周细胞的生理和功能产生了许多见解。在啮齿动物(尤其是小鼠)的研究中,基因工程动物可以表达报告蛋白,以表明周细胞的位置和生理反应参数,用于实验目的。然而,这些方法并不适用于人脑的研究。人类和啮齿类动物在生理上可能存在差异——人类的大脑与老鼠的大脑非常不同,因为人类和老鼠在大约7500万到9500万年前就开始进化分化了。
先前的大规模基因表达研究已经证实存在物种特异性的基因表达模式。即使在基因相似的小鼠品系中,血脑屏障生物学参数也存在差异。衰老的动力学(智人的平均寿命为80岁,而老鼠的平均寿命为2岁)和衰老的大脑病理是相当不同的。值得注意的是,野生型啮齿动物既不会出现阿尔茨海默病(AD)病变,也不会出现大多数其他与人类痴呆症相关的病理。
对于人类大脑周细胞的未来研究,该领域的一个重要知识缺口是缺乏普遍接受的细胞类型特异性周细胞标记物,类似于众所周知的神经元(neun和MAP2),星形胶质细胞(GFAP),小胶质细胞(IBA-1)等细胞类型特异性免疫组织化学(IHC)标记物。这种类型的细胞标记对于检测细胞在大脑中发育和存在的位置,以及它们如何响应(或作为)病理扰动而被修改非常有用。
已知在周细胞中表达的蛋白质的一个例子是血小板衍生生长因子受体β (PDGFRB),一种细胞表面酪氨酸激酶受体。功能上,PDGFRB有助于肌动蛋白细胞骨架的重排,对某些中枢神经系统功能(如脑损伤反应)至关重要。此外,AD患者周细胞中PDGFRB的缺失已被证实与血脑屏障(BBB)功能障碍有关。虽然PDGFRB在周细胞中表达,但已知其他脑细胞类型也表达PDGFRB,包括平滑肌细胞、神经上皮祖细胞和肌成纤维细胞。相应地,如下所述,研究人员知道没有PDGFRB抗体可以最佳地和特异性地描绘人脑中的周细胞。
单细胞基因组学代表了一种有前途的方法来系统地表征细胞类型特异性标记,通过测量不同细胞类型的数千个基因在单细胞分辨率下的表达水平。单细胞基因组学的最新进展大大提高了通量,可以在一次实验中对数百万个细胞进行常规分析,从而增加了大规模单细胞图谱的流行。
传统的单细胞研究可能只捕获组织中少数优势细胞类型,与之相反,更大的细胞图谱可以检测更罕见的细胞类型并识别新的细胞类型特异性标记。2023年10月9日发表在NEUROPATHOLOGY AND APPLIED NEUROBIOLOGY的文章,分析了最近生成的的scRNA-seq数据集,并确定了一种新的、敏感的和特异性的周细胞标记物,用于人类病理材料,即从福尔马林固定石蜡包埋组织块中切割的人类大脑切片。
研究人员使用一种名为EasySci的高通量和低成本的单细胞转录组测序方法,分析了来自不同人类和小鼠大脑区域的单细胞RNA表达谱。通过这项分析,我们能够识别周细胞的特定基因标记,其中一些以前没有被表征过。然后,研究人员使用商业上(因此是普遍)可用的抗体来对福尔马林固定石蜡嵌入(FFPE)人类大脑中的周细胞特异性基因产物进行免疫标记,并进行了免疫栓塞,以确定是否识别了适当大小的蛋白质。
研究结果显示,在EasySci数据集中,SLC6A12和SLC19A1具有显著的高度周细胞富集表达。针对这些蛋白质的抗体在人脑提取物的免疫块中识别出正确大小的带。在免疫组化技术优化后,两种抗体的染色在小血管中都呈强阳性,在FFPE人脑切片中染色周细胞样细胞方面,比PDGFRB抗体有效得多。在其他人类器官(肾脏、肺、肝脏、肌肉)的探索性样本中,免疫组织化学没有表现出相同的周细胞样染色模式。
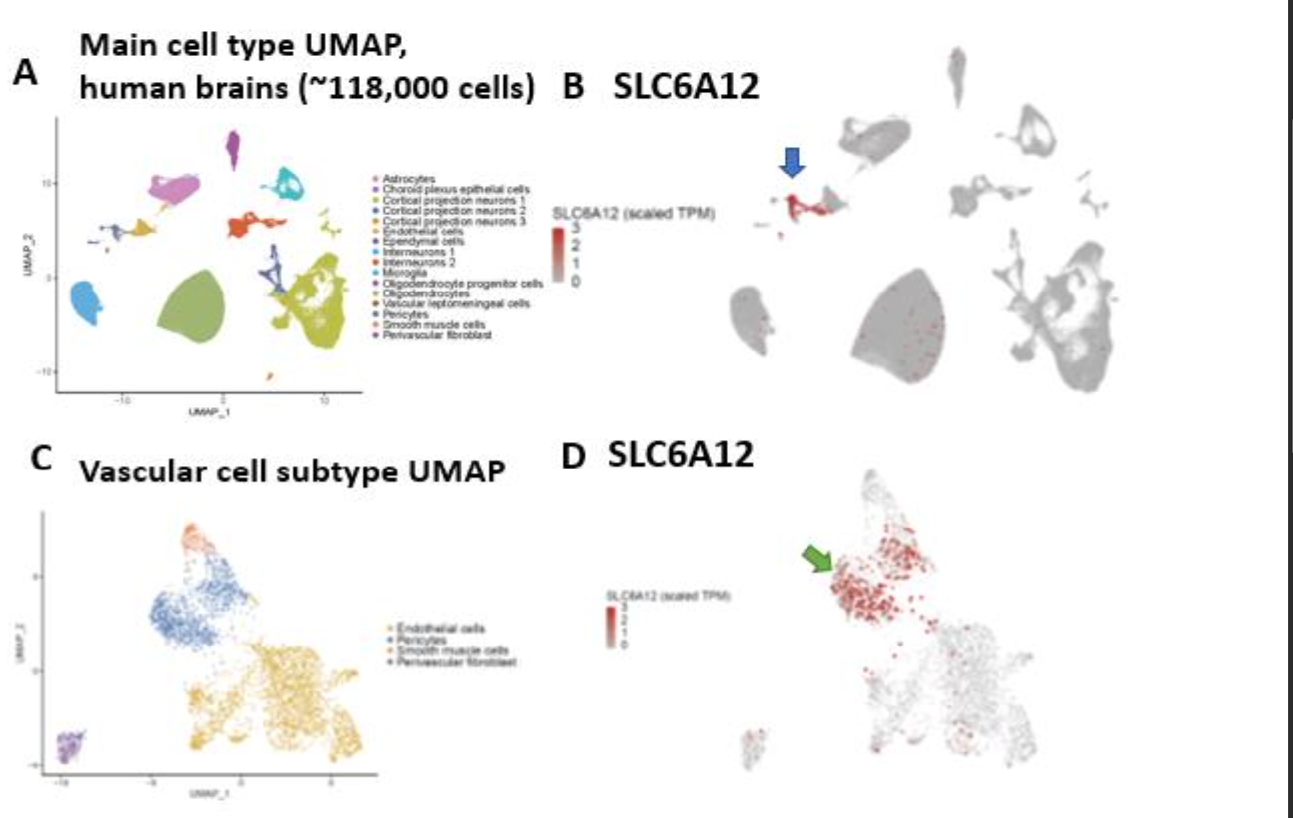
SLC6A12富含周细胞
综上所述,根据单细胞RNA-seq分析、免疫菌块和免疫组织化学研究的综合结果,SLC6A12抗体非常适合标记人类FFPE大脑切片中的周细胞。
原文出处
Sziraki, A, Zhong, Y, Neltner, AM, et al. A high-throughput single-cell RNA expression profiling method identifies human pericyte markers. Neuropathol Appl Neurobiol.e12942 2023. https://doi.org/10.1111/nan.12942


