Nat Mach Intell:细胞也能“面部识别”?AI实现纳米级精度扫描细胞异质性并识别癌细胞和病毒感染细胞
时间:2024-09-24 18:02:35 热度:37.1℃ 作者:网络
细胞表型异质性是许多生物过程的重要标志。识别细胞表型的异质性可以提供有关生物功能的关键信息,而揭示每个细胞的染色质结构则可以清楚地表征这种异质性。研究人员曾使用单分子定位显微镜(SMLM)法,特别是随机光学重建显微镜(STORM)法,来确定细胞内染色质纤维的纳米级排列,对核纳米结构的变化进行可视化和量化。STORM还能够观察到由于体细胞和干细胞的表观遗传状态不同而导致的簇相关DNA(clutch-associated DNA)的压缩变化。目前还没有报道使用SMLM图像根据亚细胞结构对细胞进行分类的技术。随着AI技术的发展,将这些精细图像用于细胞状态和类型的识别有着巨大的应用前景。
近期,西班牙巴塞罗那科学技术学院的研究团队及合作者在Nature Machine Intelligence发表了题为“A deep learning method that identifies cellular heterogeneity using nanoscale nuclear features”的文章。为了解决识别不同细胞状态的挑战,研究团队开发了一种深度学习方法——AINU(AI of the nucleus),可以在纳米级分辨率下识别特定的核特征。AINU可以根据超分辨率显微镜图像中核心组蛋白H3、RNA聚合酶II或DNA的空间排列来区分不同的细胞状态。仅用少量图像作为训练数据,AINU就能正确识别人类体细胞、人类诱导多能干细胞、转染DNA 1型单纯疱疹病毒的极早期感染细胞,甚至癌细胞。利用AI可解释性方法,研究人员发现核仁中RNA聚合酶II的定位有助于区分人类诱导多能干细胞(hiPSC)和体细胞。总之,AINU与核结构超分辨率显微镜相结合可以精确检测细胞异质性,该工具在推进再生医学、病毒学和癌症生物学的诊断和治疗方面具有相当大的潜力。

文章发表在Nature Machine Intelligence
确定超分辨率成像的最佳架构
为了选择识别体细胞和hiPSC的最佳卷积神经网络(CNN)架构及其超参数,研究人员根据独特的属性和细胞分类任务的适用性比较了11种不同的CNN架构(图1a),评估了模型在核糖体核心组蛋白H3和Pol II的总共349张细胞核双色STORM图像上的表现。研究人员使用分层五倍交叉验证方法得出了平均验证准确率,作为性能衡量标准,以80/20的训练集和验证集比例划分数据集(图1a)。在这些架构中,DenseNet-121在识别体细胞和hiPSC方面表现最佳,平均验证准确率为92.26,平均损失为0.292(图1a),因此该架构被用于后续分析。
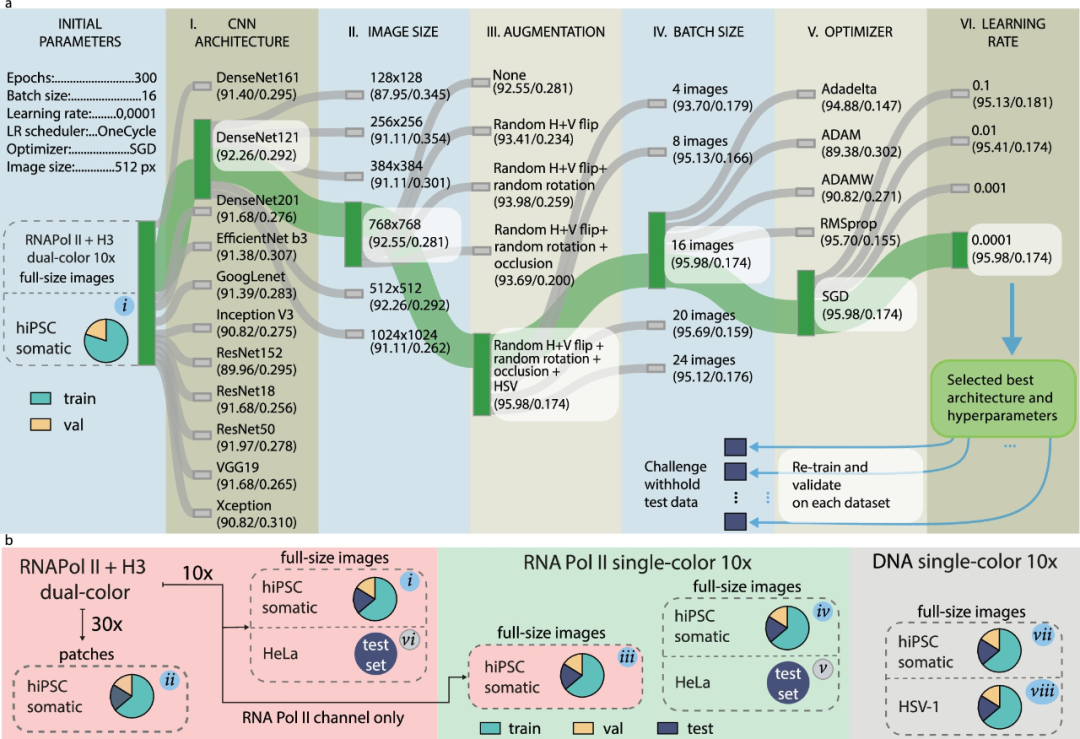
图1. 寻找AINU最佳配置的流程。
利用双色STORM图像识别hiPSC
接下来,研究人员使最佳训练配置和超参数,从头开始重新训练最佳架构,而不重复使用之前的任何权重。在重新训练阶段,整个数据集被随机划分为训练集、验证集和保留集,其中保留集占20%。为了减少随机数据集拆分或图像异质性可能造成的偏差,在保留测试集上重复进行了五次训练、验证和评估。平均而言,该模型的加权准确率和F1得分为0.85,标准偏差为0.07,操作特征(ROC)曲线下面积(AUC)为0.95 ± 0.04。在最佳数据集拆分中,模型的加权准确率和F1得分均为0.94(图2b),AUC得分为0.98(图2c),体细胞和hiPSC平均精确度和召回率分别为0.98和0.99(图2d、e)。
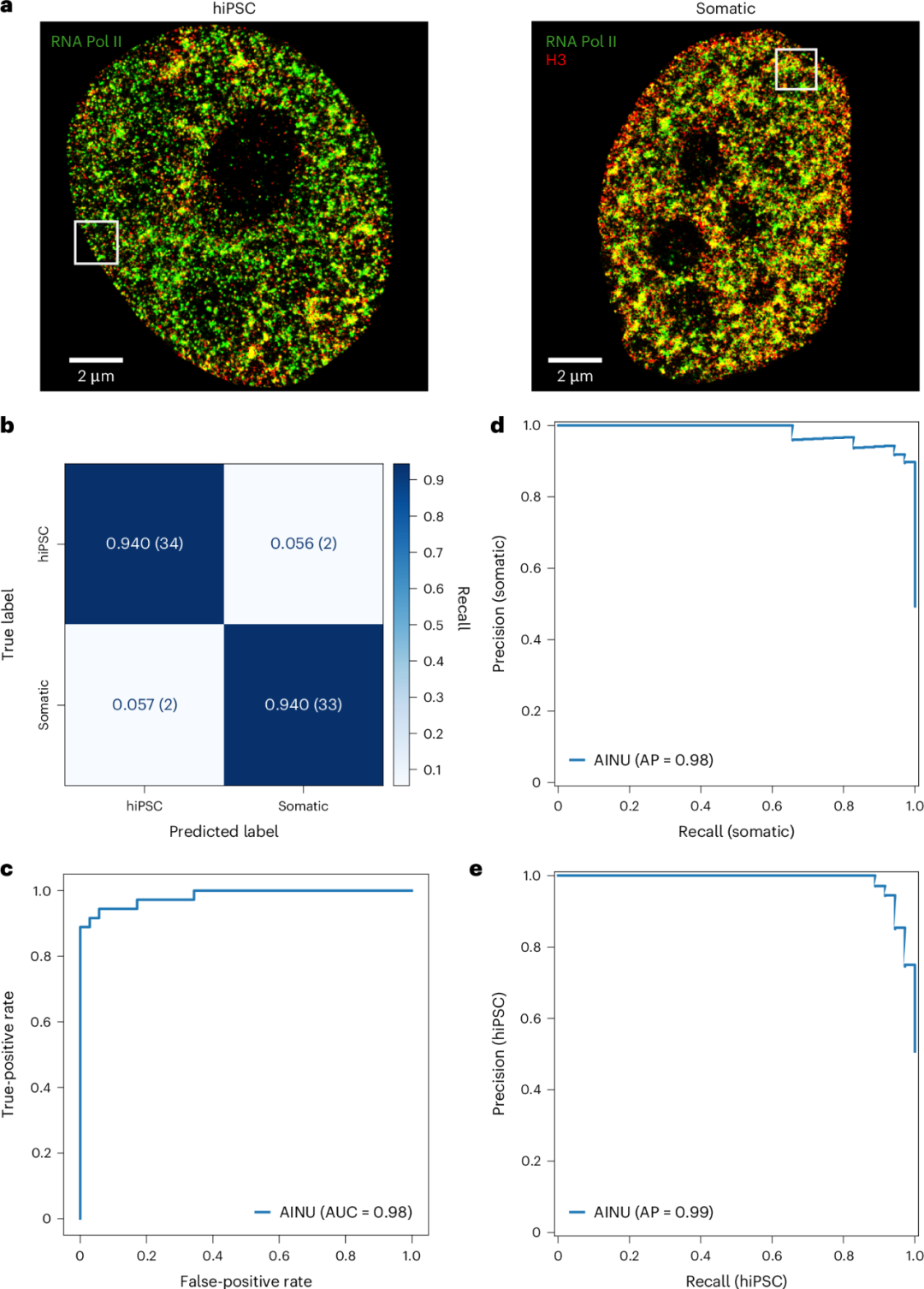
图2. 用Pol II和H3图像训练的AINU可以正确识别体细胞和hiPSC。
识别细胞状态并区分癌细胞
由于双色STORM成像既耗时又具有技术挑战性,因此研究人员测试了使用单色超分辨率(SR)成像进行训练。通过渲染双色SR定位中Pol II通道的图像生成了数据集(图3a),并添加了384份渲染自单色Pol II定位的图像(图3f),并对AINU进行了五个周期的再训练、验证和测试。结果发现,这个模型的性能与使用双色图像训练的模型非常接近。在最佳数据集拆分中,AINU识别hiPSC的准确率和F1得分为0.93,识别体细胞的准确率和F1得分为0.88(图3b),对体细胞和hiPSCs的平均准确率和召回率均为0.99(图3d、e)。此外,用hiPSC和体细胞训练的AINU模型在使用一些额外的癌细胞图像进行适当的再训练后可用于检测癌细胞,这为AINU的未来应用开辟了更多可能。
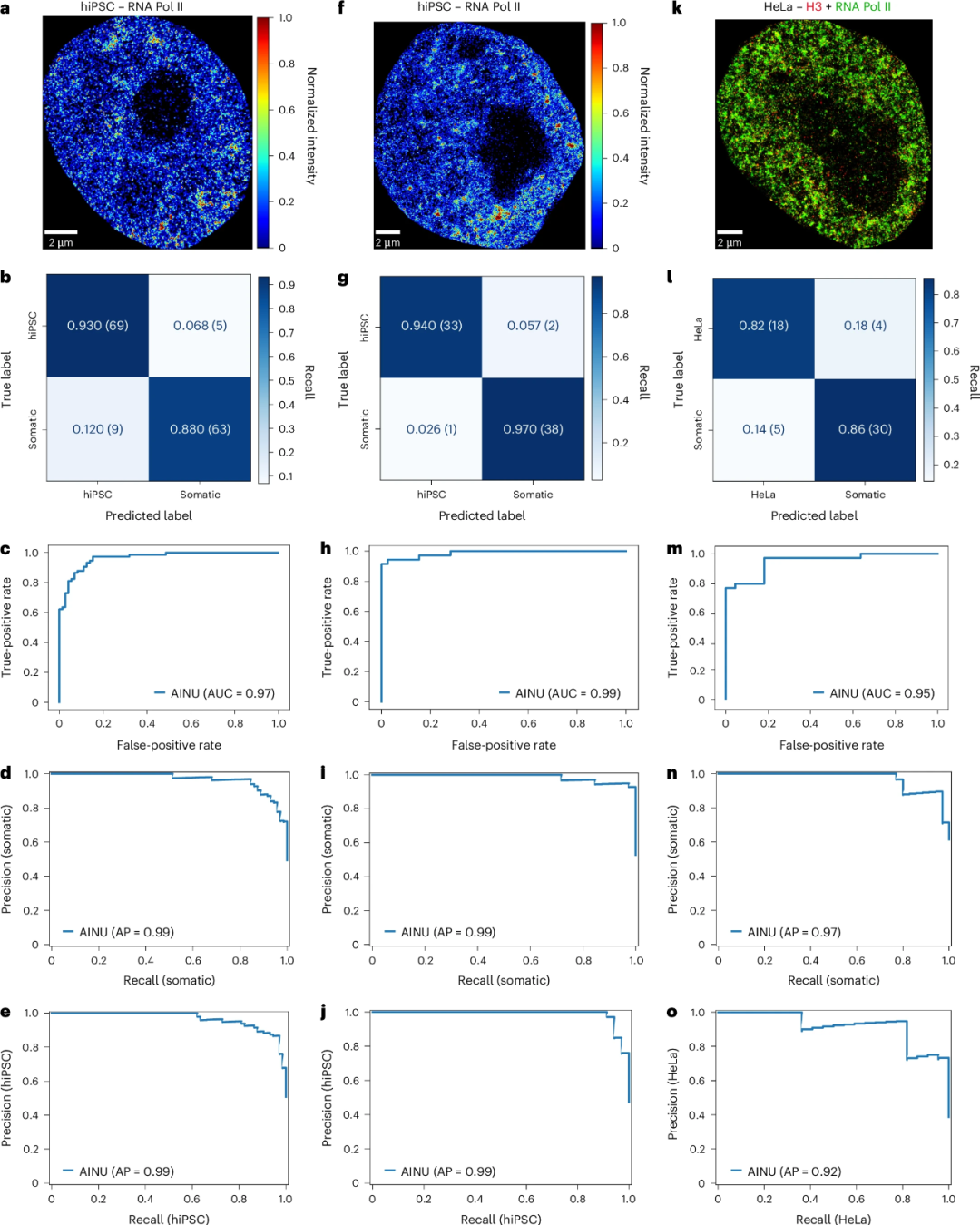
图3. 用Pol II图像训练的AINU可以正确识别体细胞和iPSC。
识别各种细胞类型
为了测试用DNA的SR图像训练的同一模型是否也能得出准确的结果,研究人员通过点击化学进行了DNA的SR成像,生成了人类体细胞(图4a)和hiPSC(图4b)的185份DNA单色SR图像数据集,分辨率提高了10倍。模型经过300次训练,对数据集进行了五次随机拆分。在保留测试集上,该模型的平均加权准确率为0.94 ± 0.04,平均AUC得分为0.98 ± 0.03。在最佳随机拆分数据集上,该模型取得了完美的表现,对hiPSC和体细胞的加权F1得分、AUC和准确率均为1(图4c)。此外,应用可解释的AI,研究人员发现AINU可以通过识别核仁Pol II(会导致asincRNA的转录增加)的存在准确地识别hiPSC。
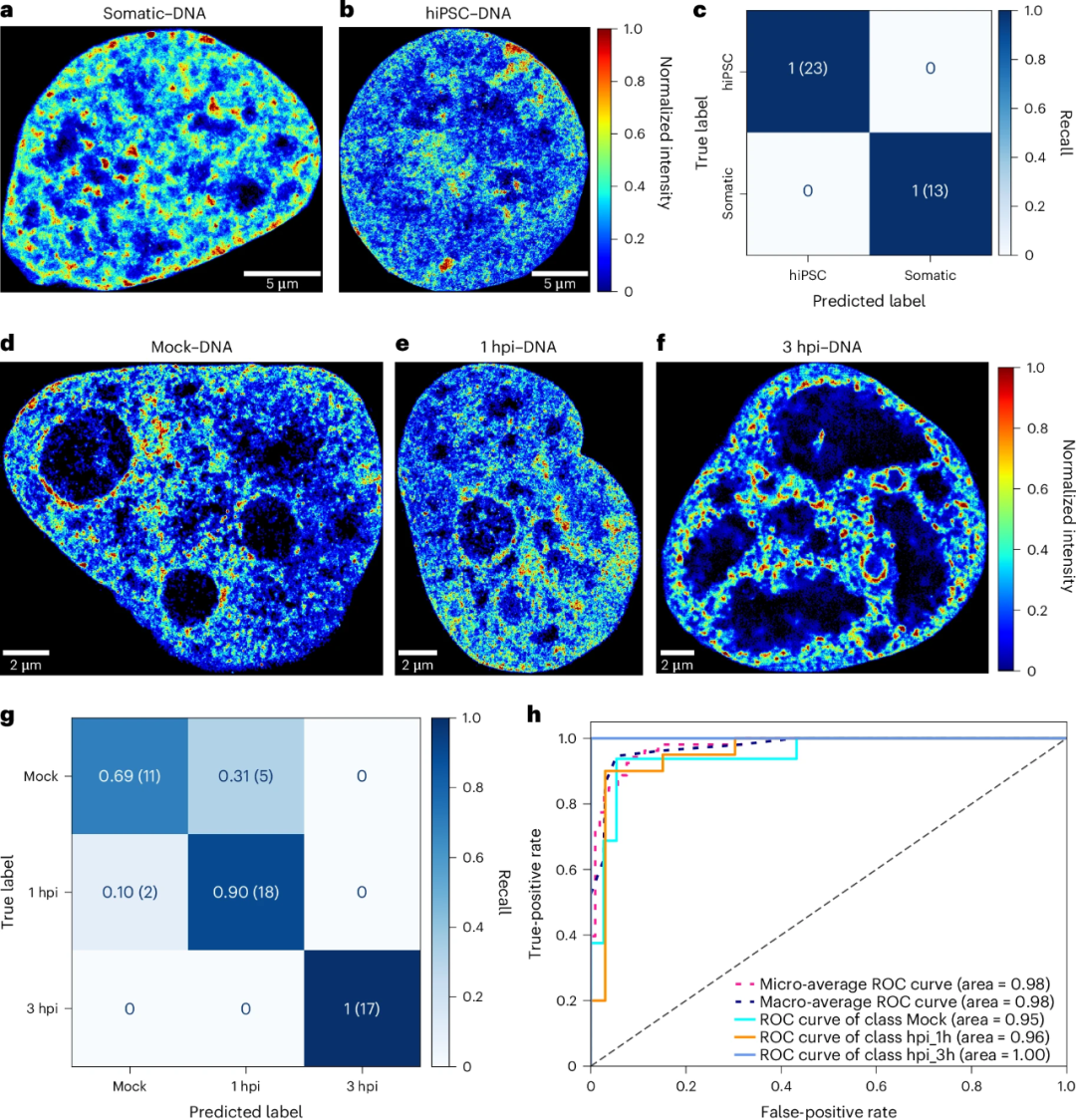
图4. 用DNA图像训练的AINU可以正确识别不同的细胞状态。
该研究表明,AINU可以识别细胞异质性并“理解”复杂的生物系统。例如,AINU只需使用免疫染色即可用于识别多能性等级较高的hiPSC克隆,而不需要繁琐的动物实验。此外,AINU还可用于在病毒感染的早期阶段识别血液或组织中的感染细胞,这对免疫学和病毒生物学有着重要的应用价值。最重要的是,AINU可以从人体样本的野生型细胞中识别癌细胞,甚至转移细胞。总而言之,AINU是通过SMLM核成像研究细胞异质性的有力工具,显示出了作为诊断方法的强大潜力。
参考资料:
https://www.nature.com/articles/s42256-024-00883-x
https://medicalxpress.com/news/2024-08-ai-cancer-viral-infections-nanoscale.html


