多伦多大学Philip M. K.小组Nat. Comput. Sci.论文:基于打分的蛋白质从头设计生成模型
时间:2023-08-17 19:38:41 热度:37.1℃ 作者:网络
深度学习为蛋白质结构预测和序列设计提供了新的方法,显著推进了蛋白质工程的发展。近期报道的蛋白序列设计模型在固定骨架设计任务中展示出强大的序列恢复能力。然而,从头生成保留活性功能位点的合理主链骨架仍然是蛋白质设计领域中的一个具有挑战性的问题。与此同时,基于连续时间评分的生成模型(Score-based generative model,SGM),也称扩散模型,已经在计算机视觉领域的2D图像生成任务中表现出优越的性能。
近日,多伦多大学Philip M. Kim小组将蛋白质骨架的从头生成任务巧妙地转化为蛋白残基间约束矩阵的图像生成问题,并提出了一个深度学习生成模型ProteinSGM,实现了蛋白质结构的精确模块化设计。该项研究工作已在Nature Computational Science期刊上发表(Nat. Comput. Sci. 2023,3, 382–392)。
ProteinSGM将输入结构的每两个残基之间Cβ-Cβ距离、ω和θ扭转角以及ϕ平面角等特征分别编码为2D的蛋白残基接触矩阵,将各矩阵拼合成6D坐标作为输入特征 (图1)。模型在6D坐标上逐步引入高斯噪音,通过学习正向扩散的过程来进行迭代训练。一旦训练完成,该模型再通过逐步去噪过程,从噪声中生成真实的6D残基约束矩阵样本。随后,使用Rosetta Design和Relax模块生成将ProteinSGM输出约束矩阵构建成相对应的蛋白质结构并进行全局能量最小化。通过这一方法,ProteinSGM能够从头产生真实的蛋白质,并将输入的蛋白骨架和功能位点修复为完整合理的蛋白结构。
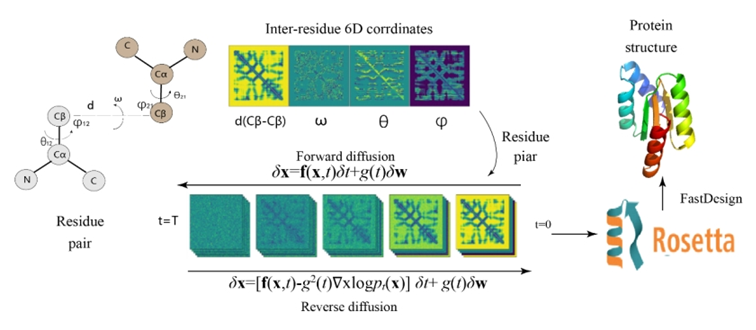
图1: ProteinSGM模型架构
研究人员对模型生成的结构进行了一系列测试。结果显示,ProteinSGM生成的结构在d、ω、θ、ϕ这些维度上与测试集的分布非常接近,这意味着该模型能够学习生成与真实蛋白质结构高度相似的6D坐标特征(图2a)。此外,作者还对6D坐标之间的联合分布进行了分析,观察到在所有的二维分布中,真实样本和生成样本之间的残基间特征具有显著的一致性(图2b)。
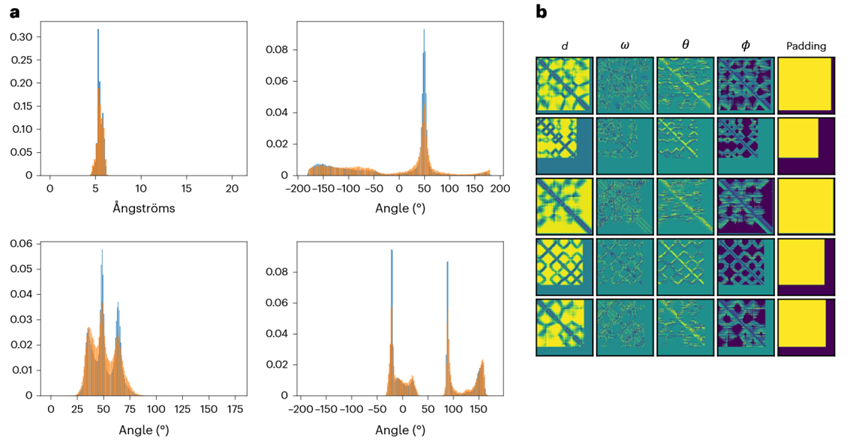
图2: ProteinSGM生成结构残基间特征分布
作者使用模型生成结构与训练集中结构的TM-score作为指标来评价模型的泛化能力和生成结构的多样性。实验发现模型生成的结构中有一部分与训练集的TM-score<0.5(图3),这表明该模型能够生成与真实蛋白质结构有所不同的全新折叠结构。
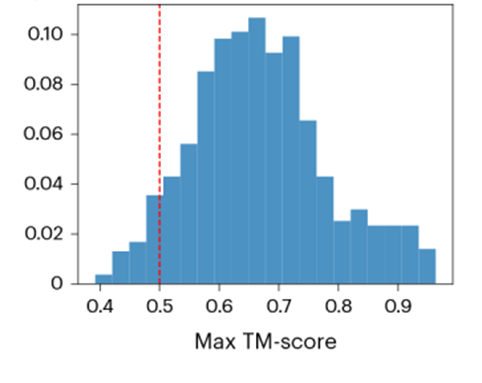
图3:ProteinSGM生成结构与测试集中结构的同源分数(TM-score)
作者对三个实际的蛋白质体系进行了ProteinSGM的条件生成功能测试。实验结果表明,在所有生成的结构中,ProteinSGM均成功地保留了指定功能区域的α-螺旋结构(图4 a,b,c中红色区域)。此外,模型还能够考虑全局结构约束,生成合适的蛋白质主链骨架结构。
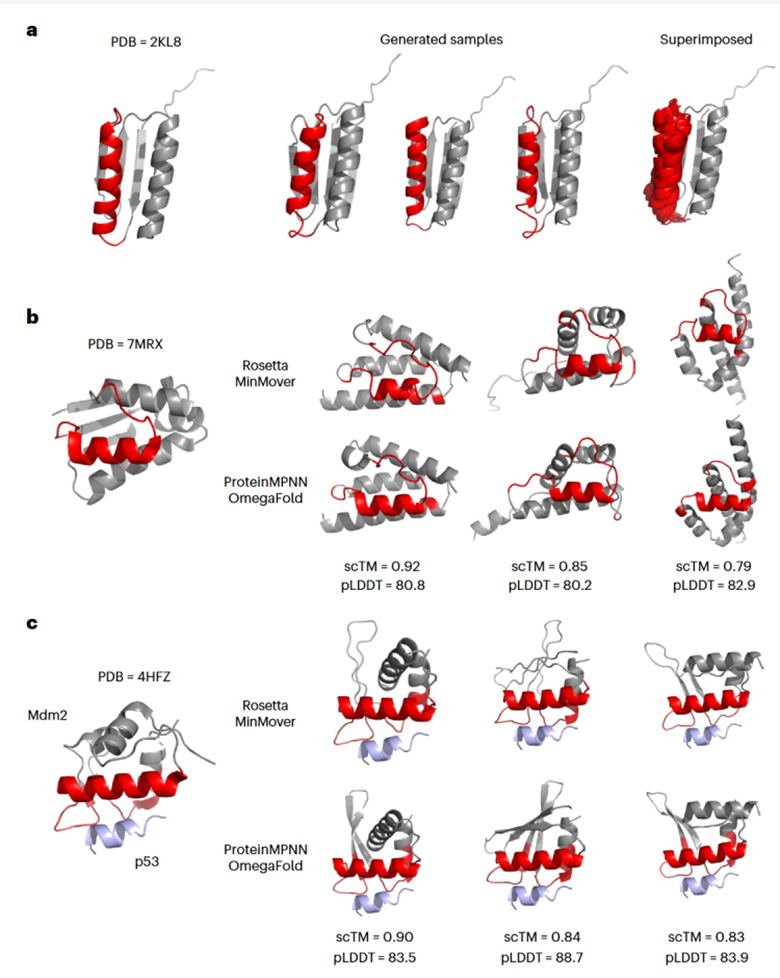
图4: ProteinSGM的条件生成实验结果展示
小结:本研究采用图像生成模型的思路,发展了一种基于蛋白质残基特征约束矩阵的3D结构生成模型ProteinSGM。通过三个真实蛋白质设计案例的测试,展现了该模型在从头生成全新蛋白质结构和精确设计特定位置的能力。然而,ProteinSGM模型在蛋白质序列长度方面存在限制,目前仅适用于单体结构生成,并需要外接结构模块将生成的2D残基约束矩阵转化为骨架结构并进行能量最小化。这一过程的计算成本较高,同时也可能伴随一定程度的特征损失。近期涌现的基于3D原子坐标的蛋白质结构扩散生成模型(如RFdiffusion等)在此方面更具优势。
参考文献
【1】Lee, J.S., Kim, J. & Kim, P.M. Score-based generative modeling for de novo protein design.Nat Comput Sci 3, 382–392 (2023).


